精准突变文库:定向进化的“高效引擎”
前言
分子定向进化是利用现代分子生物学方法,人为模拟自然进化机制,人工合成大量的突变同源基因文库。采用灵敏的定向筛选策略,创造出自然界不存在的或某些特性有显著改变的突变蛋白质或其他生物分子。
01
精准突变文库类型
精准突变文库,融合了基因合成、基因突变和定向进化研究的精华。它通过改变蛋白质的特定位点或DNA的调控区域,来研究结构和功能之间的关系。突变文库的主要类型包括:
丙氨酸扫描文库
Alanine Scan Library
实现全面突变覆盖的高级策略
利用丙氨酸体积小同时对蛋白质结构影响较小的特性,科学家通过丙氨酸系统地取代其他19个氨基酸残基的每一个,构建了丙氨酸扫描文库。这种创新方法通过探索单个氨基酸残基在蛋白质功能、活性位点和稳定性中的作用,解开了蛋白质结构和功能的秘密。

定点饱和突变文库
Site-Saturation Mutagenesis Library
实现单点氨基酸的精确突变
将蛋白序列限定区域内的氨基酸位点突变成另外的19种氨基酸,构成定点饱和突变文库。一次只突变一个氨基酸位点,每条序列和模板相比只存在一个氨基酸位点突变。这可以大幅降低引物合成成本,对于突变位点多的高库容文库极具优势。
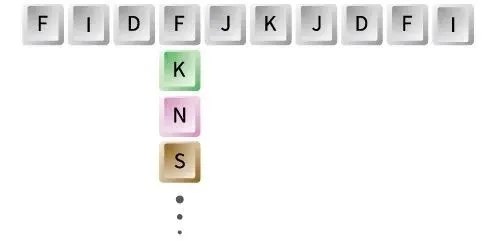
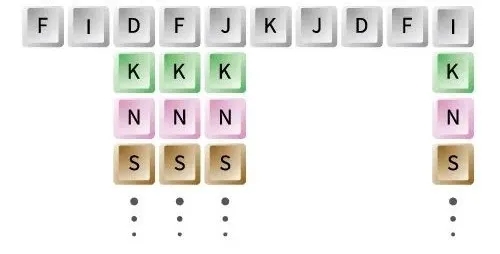
组合突变文库
Combinatorial Mutagenesis Library
实现多点多种氨基酸的精确突变
将蛋白序列的限定区域内连续的几个氨基酸位点同时突变成另外的19种氨基酸,构成组合突变文库。同时突变多个氨基酸位点,每条序列和模板相比存在多个氨基酸位点突变。为研究复杂蛋白结构与功能的关系以及进行蛋白工程、抗体研究提供了强大的实验工具。
02
精准突变文库构建流程
精确突变库旨在为研究人员提供高度精确的基因突变资源。根据设计目的不同,体外合成的精准突变文库可以更精确、更随意地控制所研究序列的多样性,有助于深入探究基因功能和调控机制。基于芯片构建精准突变文库的流程如下:
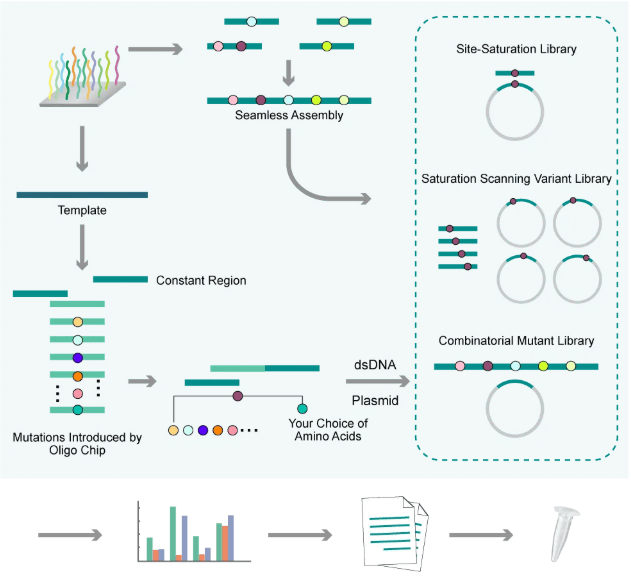
-
通过芯片引物引入突变。 -
对样品中的DNA进行大量PCR扩增。 -
将dsDNA片段无缝组装到质粒上。 -
对得到的突变文库进行测序验证及数据分析。
03
精准突变文库的应用领域
定向进化研究:构建突变文库有助于探索基因的功能和调控机制,推动基础科学研究的进展。
酶工程改造:用于改良酶的催化性能和微生物的代谢途径,为酶工程提供高效、精准的生物工具。
生物医药领域:用于筛选药物靶点的关键功能区域,发现与疾病相关的新药物靶点,促进新药开发和研究。
农业领域:有助于筛选具有改良性状的抗体、配体及受体,提高作物产量、抗病性和适应性。
食品领域:针对食品中存在的特定蛋白质进行定向进化,改善其功能性和营养价值,有助于提升食品的品质和营养水平。
Synbio Technology
合成生物学赋能技术平台
泓迅生物依托领先设计和先进制造成功交付了数百个高质量、定制库化的精准突变文库。我们根据您的需求,精心设计并合成各种DNA精准突变文库,为您提供定制化基因突变文库解决方案。
服务优势
序列控制精准
合成效率高
高度定制化
高性价比方案
泓迅案例
NNK文库是一种经典的简并突变文库,所使用的 NNK简并引物包含了32(4x4x2)种密码子组合(N= A/C/G/T, K= G/T),覆盖了所有20种氨基酸,可以将任意位点的氨基酸进行饱和突变。
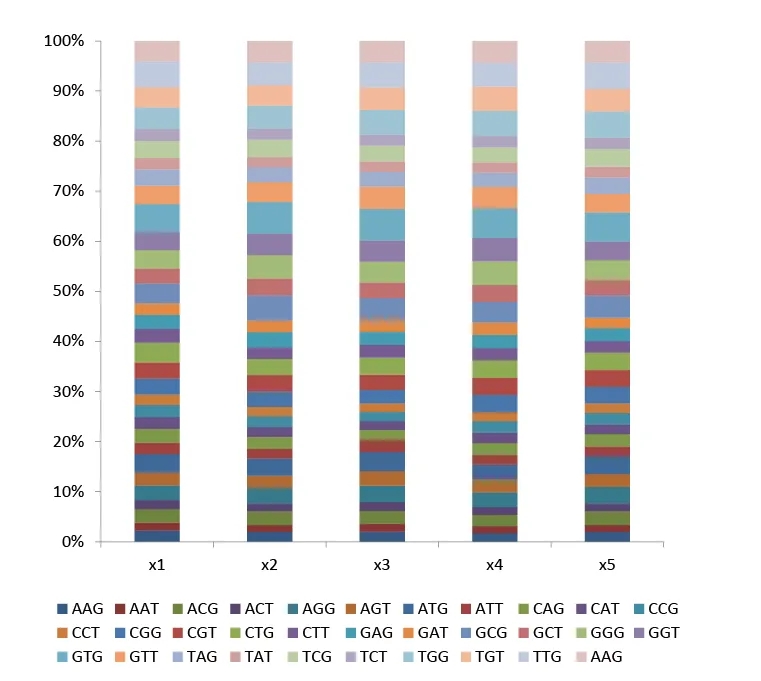
NNK文库,5个NNK位点所包含的每种密码子的比例
References
[1] Wang Y, Xue P, Cao M, Yu T, Lane ST, Zhao H. Directed Evolution: Methodologies and Applications. Chem Rev. 2021 Oct 27;121(20):12384-12444.
[2] Yu Q, Li Y, Wu B, Hu W, He M, Hu G. Novel mutagenesis and screening technologies for food microorganisms: advances and prospects. Appl Microbiol Biotechnol. 2020 Feb;104(4):1517-1531.
[3] Wang TW, Zhu H, Ma XY, Zhang T, Ma YS, Wei DZ. Mutant library construction in directed molecular evolution: casting a wider net. Mol Biotechnol. 2006 Sep;34(1):55-68.
[4] Williams G J, Domann S, Nelson A, et al. Modifying the stereochemistry of an enzyme-catalyzed reaction by directed evolution[J]. Proceedings of the National Academy of Sciences, 2003, 100(6): 3143-3148.
 Syno®C 引物合成
Syno®C 引物合成 RNA合成
RNA合成 mRNA合成
mRNA合成 Syno®GS 基因合成
Syno®GS 基因合成 载体构建
载体构建 高通量及DNA文库构建
高通量及DNA文库构建 CRISPR基因编辑平台
CRISPR基因编辑平台 病毒包装
病毒包装 基因测序及分析
基因测序及分析 重组蛋白表达平台
重组蛋白表达平台 抗体工程平台
抗体工程平台 多肽服务
多肽服务 生物信息学分析与设计
生物信息学分析与设计 CRISPR文库
CRISPR文库 合成生物学产品
合成生物学产品 ProXpress蛋白快速检测
ProXpress蛋白快速检测 CRISPR 质粒
CRISPR 质粒





















